
분석시각화 대회 코드 공유 게시물은
내용 확인 후
좋아요(투표) 가능합니다.

Semantic Segmentation을 위한 U-Net 모델 [3탄.학습 과정 특이점]
안녕하세요.
데이콘에서 활동 중인 '동화책'입니다.📚 🤓
'의미론적 분할(Semantic Segmentation)을 위한 U-Net 모델' 시리즈, 이번엔 [3탄. 훈련 과정 특이점]입니다.
읽어보시고 궁금한 사항 및 피드백이 있으시다면 언제나 편하게 댓글 달아주세요.
* 본 포스팅은 데이콘 서포터즈 "데이크루 1기" 활동의 일환입니다.
INDEX
1탄. 과제 정의 및 배경 [링크]
2탄. 모델 구조 [링크]
3탄. 학습 과정 특이점
4탄. 모델 구현 [링크]
이전 게시글에서 FCN과의 차이점을 설명하면서 U-Net은 학습 과정에서 overlap-tile과 데이터 증폭을 수행한다는 말씀을 드렸는데요
이번 게시글에서 over-tile과 데이터 증폭이 무엇이고 어떠한 장점이 있는지 정리해보겠습니다.
U-Net의 논문 제목 "U-Net: Convolutional Networks for Biomedical Image Segmentation"에서도 알 수 있듯이 U-Net은 의료 영상 이미지 분할을 위해 고안된 모델입니다. CT에서 혹의 위치를 찾거나(nodule detection) 배경으로부터 망막 혈관을 분할하는(retinal vessel segmentation) 등 병변을 진단하는데 도움이 줄 수 있습니다.
일반적인 이미지와 다르게 의료공학 분야에서는 고해상도 이미지가 대부분이기 때문에 많은 연산량을 필요로 합니다. 고용량의 의료 이미지를 효율적으로 처리하기 위한 방안으로 overlap-tile 전략을 고안해냈습니다.

그림 1. overlap-tile
그림 〈1〉은 세포 영역을 분할하는 문제를 보여주고 있습니다.
그림 〈1〉 오른쪽의 노란색 박스 영역을 분할하기 위해서는 파란색 박스 영역의 이미지 데이터가 필요합니다. 작은 영역을 예측하기 위해서 큰 영역을 학습해야한다고 이해하시면 되겠습니다.
하지만 이렇게 예측을 하게 되면 기존에 가지고 있는 원본 이미지보다 훨씬 더 작은 영역만 예측이 가능할겁니다. 하지만 우리는 입력한 전체 이미지의 분할 결과를 얻고 싶습니다.
그래서 누락되는 부분을 원본 이미지를 미러링해서 채워넣어서 전체 입력 이미지에 대해 분할할 수 있도록 하였습니다.
그림 〈1〉의 왼쪽을 자세히 보시면 원본 이미지를 둘러싼 테두리의 이미지가 원본 이미지와 거울을 댄 것처럼 대칭인 것을 확인하실 수 있습니다.
픽셀 값을 복사(미러링)함으로써 메모리의 추가적인 사용을 줄이고 원본 이미지 크기의 예측 결과를 얻어낼 수 있겠네요.

그림 2. overlap-tile (next step) [출처 : 강준영님의 vlog]
그림 〈1〉의 노란색 영역, 즉 타일을 예측하게 되면 다음 타일로 넘어가는데 필요한 영역이 이전에 예측을 위해 사용했던 영역과 겹치게(overlap) 됩니다. (그림 〈2〉)
따라서 이 방법을 overlap-tile이라고 불리게 된 것이죠.
논문에서는 overlap-tile 전략은 "GPU 메모리가 한정되어 있을 때 큰 이미지들을 인공 신경망에 적용하는데 장점이 있다"고 말하고 있습니다.
고해상도 의료 이미지에 적합한 방식이라는 생각이 드네요.
U-Net는 overlap-tile 이외에도 데이터 증폭이라는 방식을 사용하여 모델 학습을 수행합니다.
의료 공학 분야에서는 훈련할 수 있는 이미지의 갯수가 적은 반면 조직의 변이나 변형이 매우 흔하기 때문에 확보한 데이터를 증폭하는 과정이 매우 중요하다고 합니다.
데이터 증폭이란 확보한 이미지를 반전시키거나 회전, 뒤틀림, 이동시켜서 더 많은 양의 이미지를 확보하는 것을 의미합니다.
U-Net 외에도 레이블링 비용 감소 등을 위해서 다른 모델에서도 데이터 증폭은 많이 사용되고 있습니다.
또한 의료 이미지 분야에서 많은 세포들이 모여있는 경우, 즉 동일한 클래스가 인접해 있는 경우 분할하는데 많은 어려움을 겪습니다.
그림 〈3〉에서 그 예시가 나와있습니다.
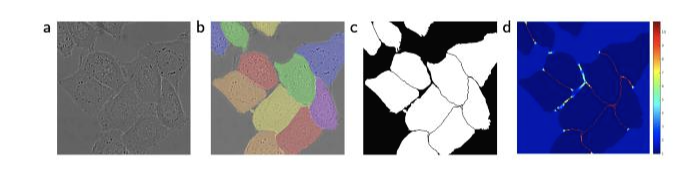
그림 3. cell segmentation
[그림 〈3〉 해석] (a)는 원본 이미지, (b)는 분할 정답을 씌어놓은 것이고 다른 색은 서로 다른 인스턴스(instance)를 의미합니다. (c)는 생성된 분할 마스크입니다. 흰색은 새포, 검정색은 배경을 의미합니다. (d)는 경계 픽셀을 학습하기 위해서 픽셀 단위로 강조된 손실함수의 가중치를 의미합니다.(아래에 더 자세히 설명)
일반적인 세포와 배경을 구분하는 것은 쉽지만 위의 예시처럼 세포가 인접해있는 경우 각각의 인스턴스(instance)를 구분하는 것은 쉽지 않습니다.
그래서 이 논문에서는 각 인스턴스의 경계와 경계 사이에 반드시 배경이 존재하도록 처리합니다. 즉 2개의 세포가 붙어있는 경우라도 둘 사이에 반드시 배경이 인식되어야하는 틈을 만들겠다는 말입니다.
이를 고려하여 손실함수를 재정의하였습니다.
weight map loss를 의미하는 항을 추가했는데 가장 가까운 세포의 경계까지의 거리와 두번째로 가까운 세포의 경계까지의 거리의 합이 최대가 되도록 하는 손실함수입니다.
이렇게 하면 모델은 낮은 손실함수를 갖는 방향으로 학습하기 때문에 두 세포 사이의 간격을 넓히는 식, 즉 두 인스턴스 사이의 배경을 넓히는 방향으로 학습하게 됩니다.
이렇게 하면 세포나 조직이 뭉쳐있는 경우에도 정확하게 인스턴스별로 분할이 가능하겠네요.
이런 의미에서 (d)에서 세포 객체들 사이에 존재하는 배경/틈에 높은 가중치가 부여된 것을 확인할 수 있습니다.
이번 게시글에서는 U-Net 훈련 상의 특이점인 overlap-tile과 데이터 증폭 그리고 재정의된 손실함수에 대해서 정리해보았습니다.
다음 게시글에서는 열심히 배운 U-Net을 실제로 구현해보도록 하겠습니다.
긴 글 읽어주셔서 감사합니다.
참고자료
정말 아이디어가 대단한 것 같습니다. 읽어주셔서 감사합니다 😀

DACON Co.,Ltd | CEO Kookjin Kim | 699-81-01021
Mail-order-sales Registration Number: 2021-서울영등포-1704
Business Providing Employment Information Number: J1204020250004
#901, Eunhaeng-ro 3, Yeongdeungpo-gu, Seoul 07237
E-mail dacon@dacon.io |
Tel. 070-4102-0545
Copyright ⓒ DACON Inc. All rights reserved
항상 다양한 모델을 접할 때 마다 손실함수를 정의해주는 것이 대단한 것 같습니다.. U-Net 모델에서 weight map loss 또한 매우 인상 깊은 것 같습니다. 좋은 정보 감사합니다 !